公開日: 09/19/2023
 多変量埋め込みの別例
多変量埋め込みの別例
この例では、多変量埋め込みの例と同じデータセットを使用し、データを2次元ではなく3次元に削減します。
1. [ヘルプ]>[サンプルデータフォルダ]を選択し、「Cytometry.jmp」を開きます。
2. [分析]>[多変量]>[多変量埋め込み]を選択します。
3. 「CD3」から「MCB」まで選択し、[Y, 列]をクリックします。
4. 「次元削減後の次元」に「3」と入力します。
5. (オプション)「手法」のコンボボックスにて[t-SNE]を選択します。また、「詳細オプション」の横のグレーの開閉アイコンをクリックし、「乱数シード値」に「1234」と入力します。
メモ: 上記のシード値を入力すると、この例で示す結果を再現できるようになります。
6. [OK]をクリックします。
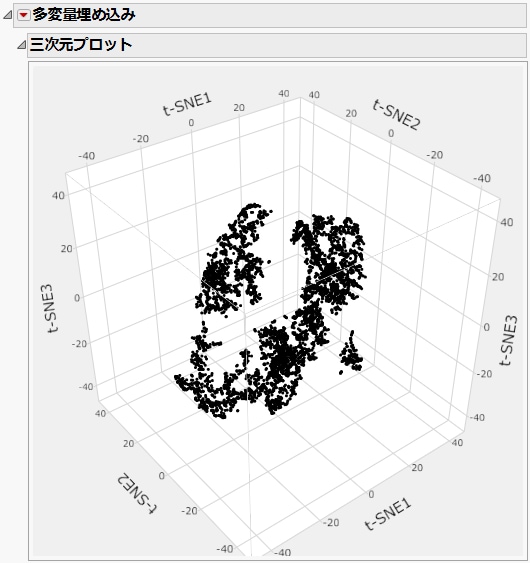
図11.4 3次元のt-SNEプロットの例
2次元への削減に比べ、3次元への削減のほうが、データがいくつかのクラスターにはっきりと分かれています。Figure 11.2の2次元の散布図に比べ、3次元のt-SNEプロットでは、クラスターがより明確になっています。
ヒント: グリッドを回転すると、はっきりと分かれているクラスターの様子を見ることができます。
より詳細な情報が必要な場合や、質問があるときは、JMPユーザーコミュニティで答えを見つけましょう (community.jmp.com).