K Meansクラスター分析の例
この例では、「K Meansクラスター分析」プラットフォームを使って、サイトメトリー(cytometry)分析データのクラスタリングを行います。
1. [ヘルプ]>[サンプルデータフォルダ]を選択し、「Cytometry.jmp」を開きます。
2. [分析]>[クラスター分析]>[K Meansクラスター分析]を選択します。
3. 「CD3」、「CD8」、「CD4」、「MCB」を選択して[Y, 列]をクリックします。
4. [OK]をクリックします。
5. 「クラスターの数」の下に「3」と入力します。
6. 「クラスター最大数(オプション)」下に「15」と入力します。
クラスターの最大個数を15に設定したため、3~15個のクラスターのあてはめが行われます。その後、これらのなかから最も適しているであろうクラスター数を選択できます。
7. [実行]をクリックします。
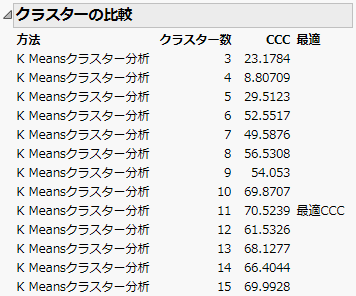
図14.2 「クラスターの比較」レポート
「クラスターの比較」レポートは、レポートウィンドウの最上部に表示されます。CCC(Cubic Clustering Criterion; 立方クラスタリング規準)の最も大きいときのクラスタリングが、最も適していると考えられるクラスター数です。この例では、クラスター数が11のときに、CCCが最大となっています。
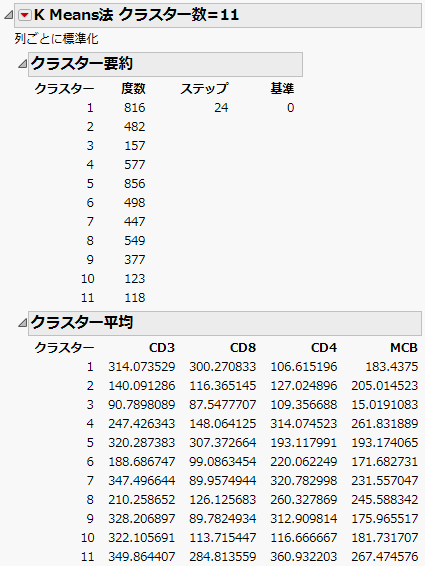
8. 「K Means法クラスター数=11」のレポートまでスクロールします。
図14.3 「K Means法クラスター数=11」レポート
「クラスター要約」レポートには、11個のクラスターそれぞれのオブザベーション数(データの行数)が表示されています。「クラスター平均」レポートには、4変数それぞれの、クラスターごとの平均が表示されます。
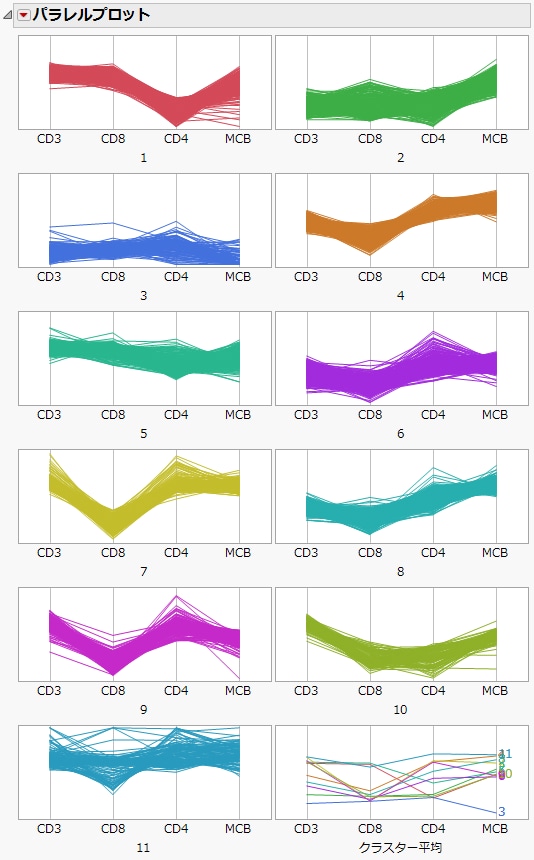
9. 「K Means法クラスター数=11」の赤い三角ボタンをクリックし、[パラレルプロット]を選択します。
図14.4 Cytometryデータのパラレルプロット
パラレルプロットからは、どのようにデータがクラスターごとに分布しているかが分かります。これらのプロットを使って、クラスター間でどのような違いがあるかを確認しましょう。クラスター4、6、7、8、9では、CD8の値が比較的低く、CD4の値が高いことがわかります。一方、クラスター1ではCD8の値が高く、CD4の値が低くなっています。
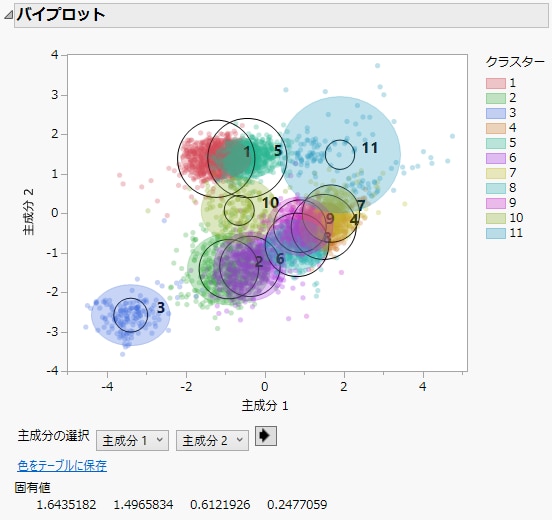
10. 「K Means法クラスター数=11」の赤い三角ボタンをクリックして[バイプロット]を選択します。
図14.5 Cytometryデータのバイプロット
プロットの右側には、クラスターを色分けする凡例が表示されます。最初の2つの主成分に基づいて、他から最も離れているように見えるクラスターは、クラスター3、10、11です。これらは、Figure 14.4のパラレルプロットを見ても、他のクラスターのプロットと異なっていることが確認できます。プロットの下のリストを使用すれば、第1主成分と第2主成分以外のバイプロットを表示できます。